Bioconductor开发的物种注释包系列集合了一个物种不同来源的注释信息,能够根据基因ID对其进行多种来源的注释,比如说基因的别名,基因的功能等。
我之前也写过一篇文章用Bioconductor对基因组注释介绍如何使用AnnotationHub下载注释数据库, 使用select(), mapIds等函数进行注释操作。我自己写一个流程也用到了它给基因ID, 如AT1G14185, 注释别名和功能描述。 注释结果中会出现一些基因无法被注释, 比如说下面这些情况, 我一直认为只是这些基因没有得到比较好的研究, 即便这些基因能够在TAIR搜到Araport的注释, 我也认为那些注释只是同源注释没有多大意义。
AT1G13970 NA NA
AT1G14120 NA NA
AT1G14240 NA NA
AT1G14600 NA NA
一开始得到的结果里没有多少个基因,所以缺少几个注释,通过手工去查找也行,但是目前差异表达分析动不动就给别人500多个基因,于是就有几十个甚至上百个未注释的基因,所以我想着要不自己更新拟南芥的物种包。
library(AnnotationHub)
ah <- AnnotationHub()
org <- ah[["AH57965"]]
org#...# TAIRGENEURL: ftp://ftp.arabidopsis.org/home/tair/Genes/TAIR10_genome_release/TAIR10_functional_descriptions#...
通过上面的代码,我找到了基因功能描述的数据库来源文件,我下载了这个文件,并且拿用AnnotationHub注释不到的功能的一个基因,”AT1G14185”,进行测试
mapIds(org, "AT1G14185", "GENENAME", "TAIR")
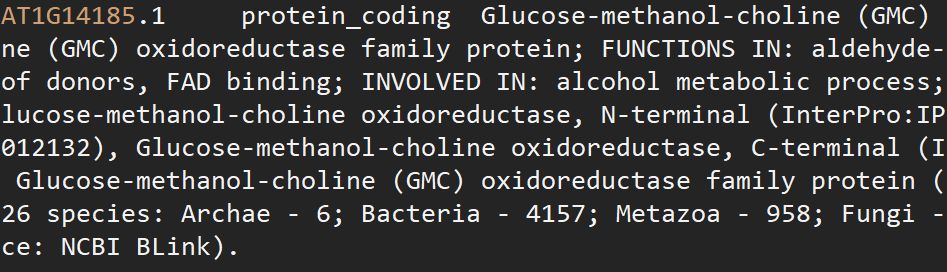
这下就非常有趣了,在原始文件中能搜索到的基因用Bioconductor的物种注释包时却没有注释信息!为了搞清楚这个原因,我花了快半个下午的时间去折腾,终于被我找到了原因。 我分别用一个能被org.At.tair.db注释和一个不能被org.At.tair.db的注释去搜索原始文本.
# 无注释
1 AT1G14185.1
2 protein_coding
3 Glucose-methanol-choline (GMC) oxidoreductase family protein
4
5 Glucose-methanol-choline (GMC) oxidoreductase family protein; FUNCTIONS IN: ...
# 有注释
1 AT1G19610.1
2 protein_coding
3 Arabidopsis defensin-like protein
4 Predicted to encode a PR (pathogenesis-related) protein. ...
5 PDF1.4; FUNCTIONS IN: molecular_function unknown;
简单的比较之后,你差不多就知道了org.At.tair.db的在功能描述这一部分其实只用第一列和第四列(为了方便展示我转置了原始数据)。这就是非常让人意外了,为啥它不用第一列和第三列呢? 我于是又去看了其他几个基因,就差不多明白了,原始的文本特别的混乱,你除了能保证第一列和第二列有信息外,其他列你根本无法保证,因此最好的策略以第一列作为检索的关键字,其他列合并成一列才行,然而作者没有那么细致。
于是我就放弃了用org.At.tair.db注释基因功能描述和基因别名了,还是自己写一个Python脚本进行注释吧。
下面这个脚本只适用于bed格式的输入,且第四列为转录本ID,另外两个输入文件分别为”gene_aliases_20140331.txt”和”TAIR10_functional_descriptions_20140331.txt”, 用法为
python bed_anno.py to_anno.bed gene_aliases_20140331.txt TAIR10_functional_descriptions_20140331.txt > anno.xls
import sysfrom collections import defaultdict
bed_file = sys.argv[1]
alias_file = sys.argv[2]
func_file = sys.argv[3]
alias_dict = defaultdict(list)
func_dict = defaultdict(list)
# read alias file
for line in open(alias_file, 'r'):
items = line.strip().split('\t')
alias_dict[items[0]] = items[1:]
# read function description file
for line in open(func_file, 'r'):
items = line.strip().split('\t')
func_dict[items[0]] = items[1:]
# annotation and output
for line in open(bed_file, 'r'):
transcript_id = line.strip().split("\t")[3]
gene_id = transcript_id.split(".")[0]
gene_alias = alias_dict[gene_id] if len(alias_dict[gene_id]) > 0 else ['']
gene_func = func_dict[transcript_id] if len(func_dict[transcript_id]) > 0 else ['']
gene_anno = '{}\t{}\t{}'.format(line.strip(), gene_alias[0], '\t'.join(gene_func))
print(gene_anno)