1、ELM训练函数elmtrain
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
|
function
[IW,B,LW,TF,TYPE] = elmtrain(P,T,N,TF,TYPE)
% ELMTRAIN Create and Train a Extreme Learning Machine
% Syntax
% [IW,B,LW,TF,TYPE] = elmtrain(P,T,N,TF,TYPE)
% Description
% Input
% P - Input Matrix of Training Set (R*Q)
% T - Output Matrix of Training Set (S*Q)
% N -
Number
of Hidden Neurons (
default
= Q)
% TF - Transfer Function:
%
'sig'
for
Sigmoidal
function
(
default
)
%
'sin'
for
Sine
function
%
'hardlim'
for
Hardlim
function
% TYPE - Regression (
0
,
default
) or Classification (
1
)
% Output
% IW - Input Weight Matrix (N*R)
% B - Bias Matrix (N*
1
)
% LW - Layer Weight Matrix (N*S)
% Example
% Regression:
% [IW,B,LW,TF,TYPE] = elmtrain(P,T,
20
,
'sig'
,
0
)
% Y = elmtrain(P,IW,B,LW,TF,TYPE)
% Classification
% [IW,B,LW,TF,TYPE] = elmtrain(P,T,
20
,
'sig'
,
1
)
% Y = elmtrain(P,IW,B,LW,TF,TYPE)
if
nargin <
2
error(
'ELM:Arguments'
,
'Not enough input arguments.'
);
end
if
nargin <
3
N = size(P,
2
);
end
if
nargin <
4
TF =
'sig'
;
end
if
nargin <
5
TYPE =
0
;
end
if
size(P,
2
) ~= size(T,
2
)
error(
'ELM:Arguments'
,
'The columns of P and T must be same.'
);
end
[R,Q] = size(P);
if
TYPE ==
1
T = ind2vec(T);
end
[S,Q] = size(T);
% Randomly Generate the Input Weight Matrix
IW = rand(N,R) *
2
-
1
;
% Randomly Generate the Bias Matrix
B = rand(N,
1
);
BiasMatrix = repmat(B,
1
,Q);
% Calculate the Layer Output Matrix H
tempH = IW * P + BiasMatrix;
switch
TF
case
'sig'
H =
1
./ (
1
+ exp(-tempH));
case
'sin'
H = sin(tempH);
case
'hardlim'
H = hardlim(tempH);
end
% Calculate the Output Weight Matrix
LW = pinv(H
') * T'
;
|
2、ELM测试函数elmtrain
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
|
function
Y = elmpredict(P,IW,B,LW,TF,TYPE)
% ELMPREDICT Simulate a Extreme Learning Machine
% Syntax
% Y = elmtrain(P,IW,B,LW,TF,TYPE)
% Description
% Input
% P - Input Matrix of Training Set (R*Q)
% IW - Input Weight Matrix (N*R)
% B - Bias Matrix (N*
1
)
% LW - Layer Weight Matrix (N*S)
% TF - Transfer Function:
%
'sig'
for
Sigmoidal
function
(
default
)
%
'sin'
for
Sine
function
%
'hardlim'
for
Hardlim
function
% TYPE - Regression (
0
,
default
) or Classification (
1
)
% Output
% Y - Simulate Output Matrix (S*Q)
% Example
% Regression:
% [IW,B,LW,TF,TYPE] = elmtrain(P,T,
20
,
'sig'
,
0
)
% Y = elmtrain(P,IW,B,LW,TF,TYPE)
% Classification
% [IW,B,LW,TF,TYPE] = elmtrain(P,T,
20
,
'sig'
,
1
)
% Y = elmtrain(P,IW,B,LW,TF,TYPE)
% See also ELMTRAIN
% Yu Lei,
11
-
7
-
2010
% Copyright www.matlabsky.com
if
nargin <
6
error(
'ELM:Arguments'
,
'Not enough input arguments.'
);
end
% Calculate the Layer Output Matrix H
Q = size(P,
2
);
BiasMatrix = repmat(B,
1
,Q);
tempH = IW * P + BiasMatrix;
switch
TF
case
'sig'
H =
1
./ (
1
+ exp(-tempH));
case
'sin'
H = sin(tempH);
case
'hardlim'
H = hardlim(tempH);
end
% Calculate the Simulate Output
Y = (H
' * LW)'
;
if
TYPE ==
1
temp_Y = zeros(size(Y));
for
i =
1
:size(Y,
2
)
[~,index] = max(Y(:,i));
temp_Y(index,i) =
1
;
end
Y = vec2ind(temp_Y);
end
|
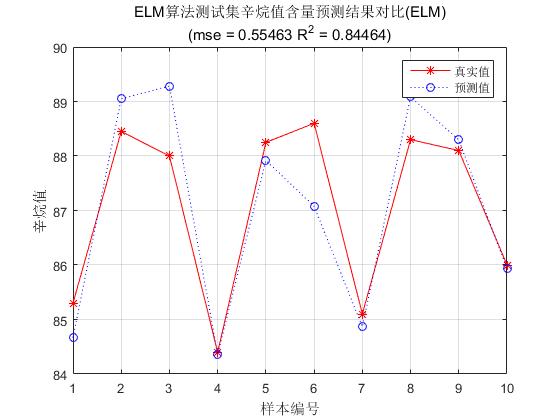
3、使用oil-dataset.mat数据集测试石油辛烷值(回归)
说明:oil_dataset.mat数据集由60个样本,401维属性组成,包含sample属性为60*401矩阵和输出label标签为60*1矩阵。
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
|
%% I. 清空环境变量
clear all
clc
%% II. 训练集/测试集产生
%%
%
1
. 导入数据
load oil_dataset.mat
%%
%
2
. 随机产生训练集和测试集
temp = randperm(size(sample,
1
));
% 训练集——
50
个样本
P_train = sample(temp(
1
:
50
),:)';
T_train =
label
(temp(
1
:
50
),:)';
% 测试集——
10
个样本
P_test = sample(temp(
51
:end),:)';
T_test =
label
(temp(
51
:end),:)';
N = size(P_test,
2
);
%% III. 数据归一化
%%
%
1
. 训练集
[p_train,inputps] = mapminmax(P_train);
p_test = mapminmax(
'apply'
,P_test,inputps);
%%
%
2
. 测试集
[t_train,outputps] = mapminmax(T_train);
t_test = mapminmax(
'apply'
,T_test,outputps);
%% IV. ELM创建/训练
[IW,B,LW,TF,TYPE] = elmtrain(p_train,t_train,
30
,
'sig'
,
0
);
%% V. ELM仿真测试
t_sim = elmpredict(p_test,IW,B,LW,TF,TYPE);
%%
%
1
. 反归一化
T_sim = mapminmax(
'reverse'
,t_sim,outputps);
%% VI. 结果对比
result = [T_test
' T_sim'
];
%%
%
1
. 均方误差
E = mse(T_sim - T_test);
%%
%
2
. 决定系数
N = length(T_test);
R2=(N*sum(T_sim.*T_test)-sum(T_sim)*sum(T_test))^
2
/((N*sum((T_sim).^
2
)-(sum(T_sim))^
2
)*(N*sum((T_test).^
2
)-(sum(T_test))^
2
));
%% VII. 绘图
figure(
1
)
plot(
1
:N,T_test,
'r-*'
,
1
:N,T_sim,
'b:o'
)
grid on
legend(
'真实值'
,
'预测值'
)
xlabel(
'样本编号'
)
ylabel(
'辛烷值'
)
string = {
'ELM算法测试集辛烷值含量预测结果对比(ELM)'
;[
'(mse = '
num2str(E)
' R^2 = '
num2str(R2)
')'
]};
title(string)
|
4、使用cement_dataset.mat数据集测试石油辛烷值(回归)
说明:数据集为混凝土抗压强度预测数据集cement_dataset.mat,包含103个样本,属性sample为:103*7矩阵,输出label为:103*1矩阵。
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
|
%数据集为混凝土抗压强度预测数据集cement_dataset.mat,将整个数据集中的
103
个样本随机划分为训练集与测试集,其中训练集包含
80
个样本,测试集包含
23
个样本;
%建立极限学习机模型,并训练;利用训练好的极限学习机模型对测试集中的
23
个样本进行预测;输出结果并绘图(真实值与预测值对比图)
%% I. 清空环境变量
clear all
clc
%% II. 训练集/测试集产生
%%
%
1
. 导入水泥样本数据,sample由
103
个样本个体组成,单个样本为
9
维属性组成,输出值在out变量中
load cement_dataset.mat
%%
%
2
. 随机产生训练集和测试集
temp = randperm(size(sample,
1
));
% 训练集——
80
个样本
P_train = sample(temp(
1
:
80
),:)';
T_train =
label
(temp(
1
:
80
),:)';
% 测试集——
23
个样本
P_test = sample(temp(
81
:end),:)';
T_test =
label
(temp(
81
:end),:)';
N = size(P_test,
2
);
%% III. 数据归一化
%%
%
1
. 训练集
[p_train,inputps] = mapminmax(P_train);
p_test = mapminmax(
'apply'
,P_test,inputps);
%%
%
2
. 测试集
[t_train,outputps] = mapminmax(T_train);
t_test = mapminmax(
'apply'
,T_test,outputps);
%% IV. ELM创建/训练
[IW,B,LW,TF,TYPE] = elmtrain(p_train,t_train,
7
,
'sig'
,
0
);
%% V. ELM仿真测试
t_sim = elmpredict(p_test,IW,B,LW,TF,TYPE);
%%
%
1
. 反归一化
T_sim = mapminmax(
'reverse'
,t_sim,outputps);
%% VI. 结果对比
result = [T_test
' T_sim'
];
%%
%
1
. 均方误差
E = mse(T_sim - T_test);
%%
%
2
. 决定系数
N = length(T_test);
R2=(N*sum(T_sim.*T_test)-sum(T_sim)*sum(T_test))^
2
/((N*sum((T_sim).^
2
)-(sum(T_sim))^
2
)*(N*sum((T_test).^
2
)-(sum(T_test))^
2
));
%% VII. 绘图
figure(
1
)
plot(
1
:N,T_test,
'r-*'
,
1
:N,T_sim,
'b:o'
)
grid on
legend(
'真实值'
,
'预测值'
)
xlabel(
'样本编号'
)
ylabel(
'水泥强度值'
)
string = {
'ELM算法测试集水泥强度值含量预测结果对比(ELM)'
;[
'(mse = '
num2str(E)
' R^2 = '
num2str(R2)
')'
]};
title(string)
|
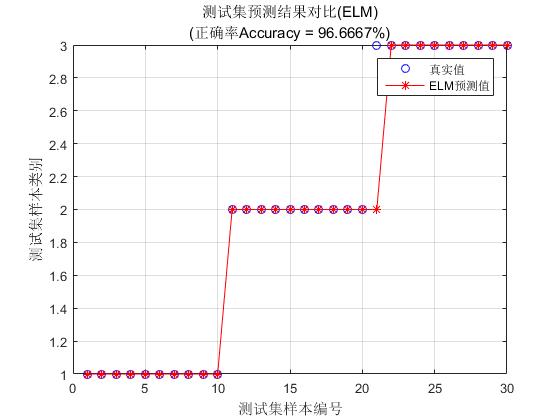
5、使用iris_dataset.mat实现分类
说明:iris以鸢尾花的特征作为数据来源,数据集包含150个数据集,分为3类,每类50个数据,每个数据包含4个属性。三类分别为:setosa, versicolor, virginica。
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
|
%% I. 清空环境变量
clear all
clc
%% II. 训练集/测试集产生
%%
%
1
. 导入数据
load iris_dataset.mat
%%
%
2
. 随机产生训练集和测试集
P_train = [];
T_train = [];
P_test = [];
T_test = [];
for
i =
1
:
3
temp_input = features((i-
1
)*
50
+
1
:i*
50
,:);
temp_output = classes((i-
1
)*
50
+
1
:i*
50
,:);
n = randperm(
50
);
% 训练集——
120
个样本
P_train = [P_train temp_input(n(
1
:
40
),:)'];
T_train = [T_train temp_output(n(
1
:
40
),:)'];
% 测试集——
30
个样本
P_test = [P_test temp_input(n(
41
:
50
),:)'];
T_test = [T_test temp_output(n(
41
:
50
),:)'];
end
%% III. ELM创建/训练
[IW,B,LW,TF,TYPE] = elmtrain(P_train,T_train,
20
,
'sig'
,
1
);
%% IV. ELM仿真测试
T_sim_1 = elmpredict(P_train,IW,B,LW,TF,TYPE);
T_sim_2 = elmpredict(P_test,IW,B,LW,TF,TYPE);
%% V. 结果对比
result_1 = [T_train
' T_sim_1'
];
result_2 = [T_test
' T_sim_2'
];
%%
%
1
. 训练集正确率
k1 = length(find(T_train == T_sim_1));
n1 = length(T_train);
Accuracy_1 = k1 / n1 *
100
;
disp([
'训练集正确率Accuracy = '
num2str(Accuracy_1)
'%('
num2str(k1)
'/'
num2str(n1)
')'
])
%%
%
2
. 测试集正确率
k2 = length(find(T_test == T_sim_2));
n2 = length(T_test);
Accuracy_2 = k2 / n2 *
100
;
disp([
'测试集正确率Accuracy = '
num2str(Accuracy_2)
'%('
num2str(k2)
'/'
num2str(n2)
')'
])
%% VI. 绘图
figure(
1
)
plot(
1
:
30
,T_test,
'bo'
,
1
:
30
,T_sim_2,
'r-*'
)
grid on
xlabel(
'测试集样本编号'
)
ylabel(
'测试集样本类别'
)
string = {
'测试集预测结果对比(ELM)'
;[
'(正确率Accuracy = '
num2str(Accuracy_2)
'%)'
]};
title(string)
legend(
'真实值'
,
'ELM预测值'
)
|
训练集正确率Accuracy = 99.1667%(119/120)
测试集正确率Accuracy = 96.6667%(29/30)
附录:官方代码(参考理解,有点啰嗦)
训练函数:
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
|
function
[TrainingTime,TrainingAccuracy] = elm_train(TrainingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
% Usage: elm_train(TrainingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
% OR: [TrainingTime, TrainingAccuracy] = elm_train(TrainingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
%
% Input:
% TrainingData_File - Filename of training data
set
% Elm_Type -
0
for
regression;
1
for
(both binary and multi-classes) classification
% NumberofHiddenNeurons -
Number
of hidden neurons assigned to the ELM
% ActivationFunction - Type of activation
function
:
%
'sig'
for
Sigmoidal
function
%
'sin'
for
Sine
function
%
'hardlim'
for
Hardlim
function
%
% Output:
% TrainingTime - Time (seconds) spent on training ELM
% TrainingAccuracy - Training accuracy:
% RMSE
for
regression or correct classification rate
for
classification
%
% MULTI-CLASSE CLASSIFICATION: NUMBER OF OUTPUT NEURONS WILL BE AUTOMATICALLY SET EQUAL TO NUMBER OF CLASSES
% FOR EXAMPLE,
if
there are
7
classes
in
all, there will have
7
output
% neurons; neuron
5
has the highest output means input belongs to
5
-th
class
%
% Sample1 regression: [TrainingTime, TrainingAccuracy, TestingAccuracy] = elm_train(
'sinc_train'
,
0
,
20
,
'sig'
)
% Sample2 classification: elm_train(
'diabetes_train'
,
1
,
20
,
'sig'
)
%
%%%% Authors: MR QIN-YU ZHU AND DR GUANG-BIN HUANG
%%%% NANYANG TECHNOLOGICAL UNIVERSITY, SINGAPORE
%%%% EMAIL: EGBHUANG@NTU.EDU.SG; GBHUANG@IEEE.ORG
%%%% WEBSITE: http:
//www.ntu.edu.sg/eee/icis/cv/egbhuang.htm
%%%% DATE: APRIL
2004
%%%%%%%%%%% Macro definition
REGRESSION=
0
;
CLASSIFIER=
1
;
%%%%%%%%%%% Load training dataset
train_data=load(TrainingData_File);
%训练集输出
T=train_data(:,
1
)';
%训练集输入
P=train_data(:,
2
:size(train_data,
2
))';
clear train_data; % Release raw training data array
%训练样本个数
NumberofTrainingData=size(P,
2
);
%输入神经元个数
NumberofInputNeurons=size(P,
1
);
%若是分类,进行处理,否则跳出
if
Elm_Type~=REGRESSION
%统计分类类别个数,并将分类结果的不同类别值存储在
label
中,
label
中元素个数即为分类的类别数
sorted_target=sort(T,
2
);
label
=zeros(
1
,
1
);
label
(
1
,
1
)=sorted_target(
1
,
1
);
j=
1
;
for
i =
2
:NumberofTrainingData
if
sorted_target(
1
,i) ~=
label
(
1
,j)
j=j+
1
;
label
(
1
,j) = sorted_target(
1
,i);
end
end
number_class=j;
NumberofOutputNeurons=number_class;
%将所有训练样本单一输出值转换为矩阵输出,例如有两类输出,则神经元输出结果为[
1
;
0
]或[
0
;
1
]
temp_T=zeros(NumberofOutputNeurons, NumberofTrainingData);
for
i =
1
:NumberofTrainingData
for
j =
1
:number_class
if
label
(
1
,j) == T(
1
,i)
break
;
end
end
temp_T(j,i)=
1
;
end
T=temp_T*
2
-
1
;
end
% Calculate weights & biases
start_time_train=cputime;
% Random generate input weights InputWeight (w_i) and biases BiasofHiddenNeurons (b_i) of hidden neurons
InputWeight=rand(NumberofHiddenNeurons,NumberofInputNeurons)*
2
-
1
;
BiasofHiddenNeurons=rand(NumberofHiddenNeurons,
1
);
tempH=InputWeight*P;
clear P; % Release input of training data
ind=ones(
1
,NumberofTrainingData);
BiasMatrix=BiasofHiddenNeurons(:,ind); % Extend the bias matrix BiasofHiddenNeurons to match the demention of H
tempH=tempH+BiasMatrix;
%%%%%%%%%%% Calculate hidden neuron output matrix H
switch
lower(ActivationFunction)
case
{
'sig'
,
'sigmoid'
}
%%%%%%%% Sigmoid
H =
1
./ (
1
+ exp(-tempH));
case
{
'sin'
,
'sine'
}
%%%%%%%% Sine
H = sin(tempH);
case
{
'hardlim'
}
%%%%%%%% Hard Limit
H = hardlim(tempH);
%%%%%%%% More activation functions can be added here
end
clear tempH; % Release the temparary array
for
calculation of hidden neuron output matrix H
%%%%%%%%%%% Calculate output weights OutputWeight (beta_i)
OutputWeight=pinv(H
') * T'
;
end_time_train=cputime;
TrainingTime=end_time_train-start_time_train % Calculate CPU time (seconds) spent
for
training ELM
%%%%%%%%%%% Calculate the training accuracy
Y=(H
' * OutputWeight)'
; % Y: the actual output of the training data
if
Elm_Type == REGRESSION
TrainingAccuracy=sqrt(mse(T - Y)) % Calculate training accuracy (RMSE)
for
regression
case
output=Y;
end
clear H;
if
Elm_Type == CLASSIFIER
%%%%%%%%%% Calculate training & testing classification accuracy
MissClassificationRate_Training=
0
;
for
i =
1
: size(T,
2
)
[x, label_index_expected]=max(T(:,i));
[x, label_index_actual]=max(Y(:,i));
output(i)=
label
(label_index_actual);
if
label_index_actual~=label_index_expected
MissClassificationRate_Training=MissClassificationRate_Training+
1
;
end
end
TrainingAccuracy=
1
-MissClassificationRate_Training/NumberofTrainingData
end
if
Elm_Type~=REGRESSION
save(
'elm_model'
,
'NumberofInputNeurons'
,
'NumberofOutputNeurons'
,
'InputWeight'
,
'BiasofHiddenNeurons'
,
'OutputWeight'
,
'ActivationFunction'
,
'label'
,
'Elm_Type'
);
else
save(
'elm_model'
,
'InputWeight'
,
'BiasofHiddenNeurons'
,
'OutputWeight'
,
'ActivationFunction'
,
'Elm_Type'
);
end
|
测试函数:
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
|
function
[TestingTime, TestingAccuracy] = elm_predict(TestingData_File)
% Usage: elm_predict(TestingData_File)
% OR: [TestingTime, TestingAccuracy] = elm_predict(TestingData_File)
%
% Input:
% TestingData_File - Filename of testing data
set
%
% Output:
% TestingTime - Time (seconds) spent on predicting ALL testing data
% TestingAccuracy - Testing accuracy:
% RMSE
for
regression or correct classification rate
for
classification
%
% MULTI-CLASSE CLASSIFICATION: NUMBER OF OUTPUT NEURONS WILL BE AUTOMATICALLY SET EQUAL TO NUMBER OF CLASSES
% FOR EXAMPLE,
if
there are
7
classes
in
all, there will have
7
output
% neurons; neuron
5
has the highest output means input belongs to
5
-th
class
%
% Sample1 regression: [TestingTime, TestingAccuracy] = elm_predict(
'sinc_test'
)
% Sample2 classification: elm_predict(
'diabetes_test'
)
%
%%%% Authors: MR QIN-YU ZHU AND DR GUANG-BIN HUANG
%%%% NANYANG TECHNOLOGICAL UNIVERSITY, SINGAPORE
%%%% EMAIL: EGBHUANG@NTU.EDU.SG; GBHUANG@IEEE.ORG
%%%% WEBSITE: http:
//www.ntu.edu.sg/eee/icis/cv/egbhuang.htm
%%%% DATE: APRIL
2004
%%%%%%%%%%% Macro definition
REGRESSION=
0
;
CLASSIFIER=
1
;
%%%%%%%%%%% Load testing dataset
test_data=load(TestingData_File);
TV.T=test_data(:,
1
)';
TV.P=test_data(:,
2
:size(test_data,
2
))';
clear test_data; % Release raw testing data array
NumberofTestingData=size(TV.P,
2
);
load elm_model.mat;
if
Elm_Type~=REGRESSION
%%%%%%%%%% Processing the targets of testing
temp_TV_T=zeros(NumberofOutputNeurons, NumberofTestingData);
for
i =
1
:NumberofTestingData
for
j =
1
:size(
label
,
2
)
if
label
(
1
,j) == TV.T(
1
,i)
break
;
end
end
temp_TV_T(j,i)=
1
;
end
TV.T=temp_TV_T*
2
-
1
;
end % end
if
of Elm_Type
%%%%%%%%%%% Calculate the output of testing input
start_time_test=cputime;
tempH_test=InputWeight*TV.P;
clear TV.P; % Release input of testing data
ind=ones(
1
,NumberofTestingData);
BiasMatrix=BiasofHiddenNeurons(:,ind); % Extend the bias matrix BiasofHiddenNeurons to match the demention of H
tempH_test=tempH_test + BiasMatrix;
switch
lower(ActivationFunction)
case
{
'sig'
,
'sigmoid'
}
%%%%%%%% Sigmoid
H_test =
1
./ (
1
+ exp(-tempH_test));
case
{
'sin'
,
'sine'
}
%%%%%%%% Sine
H_test = sin(tempH_test);
case
{
'hardlim'
}
%%%%%%%% Hard Limit
H_test = hardlim(tempH_test);
%%%%%%%% More activation functions can be added here
end
TY=(H_test
' * OutputWeight)'
; % TY: the actual output of the testing data
end_time_test=cputime;
TestingTime=end_time_test-start_time_test % Calculate CPU time (seconds) spent by ELM predicting the whole testing data
if
Elm_Type == REGRESSION
TestingAccuracy=sqrt(mse(TV.T - TY)) % Calculate testing accuracy (RMSE)
for
regression
case
output=TY;
end
if
Elm_Type == CLASSIFIER
%%%%%%%%%% Calculate training & testing classification accuracy
MissClassificationRate_Testing=
0
;
for
i =
1
: size(TV.T,
2
)
[x, label_index_expected]=max(TV.T(:,i));
[x, label_index_actual]=max(TY(:,i));
output(i)=
label
(label_index_actual);
if
label_index_actual~=label_index_expected
MissClassificationRate_Testing=MissClassificationRate_Testing+
1
;
end
end
TestingAccuracy=
1
-MissClassificationRate_Testing/NumberofTestingData
end
save(
'elm_output'
,
'output'
);
|
训练和测试在一起:
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
|
function
[TrainingTime, TestingTime, TrainingAccuracy, TestingAccuracy] = elm(TrainingData_File, TestingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
% Usage: elm(TrainingData_File, TestingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
% OR: [TrainingTime, TestingTime, TrainingAccuracy, TestingAccuracy] = elm(TrainingData_File, TestingData_File, Elm_Type, NumberofHiddenNeurons, ActivationFunction)
%
% Input:
% TrainingData_File - Filename of training data
set
% TestingData_File - Filename of testing data
set
% Elm_Type -
0
for
regression;
1
for
(both binary and multi-classes) classification
% NumberofHiddenNeurons -
Number
of hidden neurons assigned to the ELM
% ActivationFunction - Type of activation
function
:
%
'sig'
for
Sigmoidal
function
%
'sin'
for
Sine
function
%
'hardlim'
for
Hardlim
function
%
'tribas'
for
Triangular basis
function
%
'radbas'
for
Radial basis
function
(
for
additive type of SLFNs instead of RBF type of SLFNs)
%
% Output:
% TrainingTime - Time (seconds) spent on training ELM
% TestingTime - Time (seconds) spent on predicting ALL testing data
% TrainingAccuracy - Training accuracy:
% RMSE
for
regression or correct classification rate
for
classification
% TestingAccuracy - Testing accuracy:
% RMSE
for
regression or correct classification rate
for
classification
%
% MULTI-CLASSE CLASSIFICATION: NUMBER OF OUTPUT NEURONS WILL BE AUTOMATICALLY SET EQUAL TO NUMBER OF CLASSES
% FOR EXAMPLE,
if
there are
7
classes
in
all, there will have
7
output
% neurons; neuron
5
has the highest output means input belongs to
5
-th
class
%
% Sample1 regression: [TrainingTime, TestingTime, TrainingAccuracy, TestingAccuracy] = ELM(
'sinc_train'
,
'sinc_test'
,
0
,
20
,
'sig'
)
% Sample2 classification: elm(
'diabetes_train'
,
'diabetes_test'
,
1
,
20
,
'sig'
)
%
%%%% Authors: MR QIN-YU ZHU AND DR GUANG-BIN HUANG
%%%% NANYANG TECHNOLOGICAL UNIVERSITY, SINGAPORE
%%%% EMAIL: EGBHUANG@NTU.EDU.SG; GBHUANG@IEEE.ORG
%%%% WEBSITE: http:
//www.ntu.edu.sg/eee/icis/cv/egbhuang.htm
%%%% DATE: APRIL
2004
%%%%%%%%%%% Macro definition
REGRESSION=
0
;
CLASSIFIER=
1
;
%%%%%%%%%%% Load training dataset
train_data=load(TrainingData_File);
%训练集输出
T=train_data(:,
1
)';
%训练集输入
P=train_data(:,
2
:size(train_data,
2
))';
clear train_data; % Release raw training data array
%%%%%%%%%%% Load testing dataset
test_data=load(TestingData_File);
%测试集输出
TV.T=test_data(:,
1
)';
%测试集输入
TV.P=test_data(:,
2
:size(test_data,
2
))';
clear test_data; % Release raw testing data array
%训练样本个数
NumberofTrainingData=size(P,
2
);
%测试样本个数
NumberofTestingData=size(TV.P,
2
);
%输入神经元个数
NumberofInputNeurons=size(P,
1
);
%若是分类,进行处理,否则跳出
if
Elm_Type~=REGRESSION
%统计分类类别个数,并将分类结果的不同类别值存储在
label
中,
label
中元素个数即为分类的类别数
sorted_target=sort(cat(
2
,T,TV.T),
2
);
label
=zeros(
1
,
1
); % Find and save
in
'label'
class
label
from training and testing data sets
label
(
1
,
1
)=sorted_target(
1
,
1
);
j=
1
;
for
i =
2
:(NumberofTrainingData+NumberofTestingData)
if
sorted_target(
1
,i) ~=
label
(
1
,j)
j=j+
1
;
label
(
1
,j) = sorted_target(
1
,i);
end
end
number_class=j;
NumberofOutputNeurons=number_class;
%将所有训练样本单一输出值转换为矩阵输出,例如有两类输出,则神经元输出结果为[
1
;
0
]或[
0
;
1
]
temp_T=zeros(NumberofOutputNeurons, NumberofTrainingData);
for
i =
1
:NumberofTrainingData
for
j =
1
:number_class
if
label
(
1
,j) == T(
1
,i)
break
;
end
end
temp_T(j,i)=
1
;
end
%将神经元输出值转换为(-
1
,
1
)之间
T=temp_T*
2
-
1
;
%T=temp_T;
%将所有测试样本单一输出值转换为矩阵输出,例如有两类输出,则神经元输出结果为[
1
;
0
]或[
0
;
1
]
temp_TV_T=zeros(NumberofOutputNeurons, NumberofTestingData);
for
i =
1
:NumberofTestingData
for
j =
1
:number_class
if
label
(
1
,j) == TV.T(
1
,i)
break
;
end
end
temp_TV_T(j,i)=
1
;
end
TV.T=temp_TV_T*
2
-
1
;
%TV.T=temp_TV_T;
end
%结束分类
%Calculate weights & biases
start_time_train=cputime;
%%%%%%%%%%% Random generate input weights InputWeight (w_i) and biases BiasofHiddenNeurons (b_i) of hidden neurons
InputWeight=rand(NumberofHiddenNeurons,NumberofInputNeurons)*
2
-
1
;
BiasofHiddenNeurons=rand(NumberofHiddenNeurons,
1
);
tempH=InputWeight*P;
clear P; % Release input of training data
ind=ones(
1
,NumberofTrainingData);
BiasMatrix=BiasofHiddenNeurons(:,ind); % Extend the bias matrix BiasofHiddenNeurons to match the demention of H
tempH=tempH+BiasMatrix;
%%%%%%%%%%% Calculate hidden neuron output matrix H
switch
lower(ActivationFunction)
case
{
'sig'
,
'sigmoid'
}
%%%%%%%% Sigmoid
H =
1
./ (
1
+ exp(-tempH));
case
{
'sin'
,
'sine'
}
%%%%%%%% Sine
H = sin(tempH);
case
{
'hardlim'
}
%%%%%%%% Hard Limit
H = double(hardlim(tempH));
case
{
'tribas'
}
%%%%%%%% Triangular basis
function
H = tribas(tempH);
case
{
'radbas'
}
%%%%%%%% Radial basis
function
H = radbas(tempH);
%%%%%%%% More activation functions can be added here
end
clear tempH; % Release the temparary array
for
calculation of hidden neuron output matrix H
%%%%%%%%%%% Calculate output weights OutputWeight (beta_i)
OutputWeight=pinv(H
') * T'
; % implementation without regularization factor
//refer to 2006 Neurocomputing paper
%OutputWeight=inv(eye(size(H,
1
))/C+H * H
') * H * T'
; % faster method
1
//refer to 2012 IEEE TSMC-B paper
%implementation; one can
set
regularizaiton factor C properly
in
classification applications
%OutputWeight=(eye(size(H,
1
))/C+H * H
') \ H * T'
; % faster method
2
//refer to 2012 IEEE TSMC-B paper
%implementation; one can
set
regularizaiton factor C properly
in
classification applications
%If you
use
faster methods or kernel method, PLEASE CITE
in
your paper properly:
%Guang-Bin Huang, Hongming Zhou, Xiaojian Ding, and Rui Zhang,
"Extreme Learning Machine for Regression and Multi-Class Classification,"
submitted to IEEE Transactions on Pattern Analysis and Machine Intelligence, October
2010
.
end_time_train=cputime;
TrainingTime=end_time_train-start_time_train % Calculate CPU time (seconds) spent
for
training ELM
%%%%%%%%%%% Calculate the training accuracy
Y=(H
' * OutputWeight)'
; % Y: the actual output of the training data
if
Elm_Type == REGRESSION
TrainingAccuracy=sqrt(mse(T - Y)) % Calculate training accuracy (RMSE)
for
regression
case
end
clear H;
%%%%%%%%%%% Calculate the output of testing input
start_time_test=cputime;
tempH_test=InputWeight*TV.P;
clear TV.P; % Release input of testing data
ind=ones(
1
,NumberofTestingData);
BiasMatrix=BiasofHiddenNeurons(:,ind); % Extend the bias matrix BiasofHiddenNeurons to match the demention of H
tempH_test=tempH_test + BiasMatrix;
switch
lower(ActivationFunction)
case
{
'sig'
,
'sigmoid'
}
%%%%%%%% Sigmoid
H_test =
1
./ (
1
+ exp(-tempH_test));
case
{
'sin'
,
'sine'
}
%%%%%%%% Sine
H_test = sin(tempH_test);
case
{
'hardlim'
}
%%%%%%%% Hard Limit
H_test = hardlim(tempH_test);
case
{
'tribas'
}
%%%%%%%% Triangular basis
function
H_test = tribas(tempH_test);
case
{
'radbas'
}
%%%%%%%% Radial basis
function
H_test = radbas(tempH_test);
%%%%%%%% More activation functions can be added here
end
TY=(H_test
' * OutputWeight)'
; % TY: the actual output of the testing data
end_time_test=cputime;
TestingTime=end_time_test-start_time_test % Calculate CPU time (seconds) spent by ELM predicting the whole testing data
if
Elm_Type == REGRESSION
TestingAccuracy=sqrt(mse(TV.T - TY)) % Calculate testing accuracy (RMSE)
for
regression
case
end
if
Elm_Type == CLASSIFIER
%%%%%%%%%% Calculate training & testing classification accuracy
MissClassificationRate_Training=
0
;
MissClassificationRate_Testing=
0
;
for
i =
1
: size(T,
2
)
[x, label_index_expected]=max(T(:,i));
[x, label_index_actual]=max(Y(:,i));
if
label_index_actual~=label_index_expected
MissClassificationRate_Training=MissClassificationRate_Training+
1
;
end
end
TrainingAccuracy=
1
-MissClassificationRate_Training/size(T,
2
)
for
i =
1
: size(TV.T,
2
)
[x, label_index_expected]=max(TV.T(:,i));
[x, label_index_actual]=max(TY(:,i));
if
label_index_actual~=label_index_expected
MissClassificationRate_Testing=MissClassificationRate_Testing+
1
;
end
end
TestingAccuracy=
1
-MissClassificationRate_Testing/size(TV.T,
2
)
end
|
附件:http://down.51cto.com/data/2367654
本文转自stock0991 51CTO博客,原文链接:http://blog.51cto.com/qing0991/1769248,如需转载请自行联系原作者