新智元报道
来源:HIN
作者:大明,闻菲
【新智元导读】NIH临床中心最新公布了一个迄今规模最大的多类别、病灶级别标注临床医疗CT图像开放数据集DeepLesion,研究人员在此基础上训练深度神经网络,创建了一个具有统一框架的大规模通用病灶检测器,能够更准确、更自动地衡量患者体内所有病灶的大小,实现全身范围的癌症初步评估。

- 4427名独立的匿名患者
- 10594次CT扫描(平均每位患者有3次随访)
- 32735个带标记的病灶实例
- 一共928020张CT横切图像(512×512分辨率)
这就是美国国家卫生研究院(NIH)临床中心最新公开发布的大型CT图像数据集DeepLesion,也是迄今全球规模最大的多类别、病灶级别标注的开放获取临床医疗图像数据集。
“我们希望这项工作能更好地帮助放射科医生进行诊断,解决真正有意义的临床问题。”DeepLesion项目技术负责人、现在已经离开NIH、出任平安科技美国东部研究院院长的吕乐博士在接受新智元采访时说:“这件工作首先离不开我们在NIH的临床医生合作者罗纳德·撒莫斯(Ronald M. Summers),他是医生里面对技术理解特别深刻的,为获取这个临床的大规模医疗数据集提供了极大的帮助和指导。”
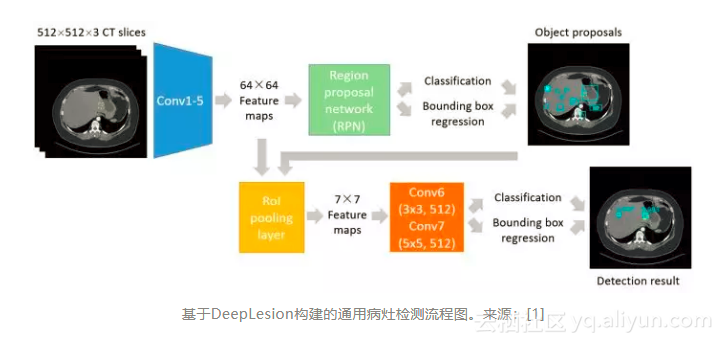
“基于DeepLesion数据集,我们开发了一种通用的病变检测器,第一次为帮助放射科医生找到患者身上所有类型的病灶提供了技术可能。”DeepLesion项目的主要完成人,NIH临床中心的博士后研究员闫轲介绍说。
目前,大多数公开可用的医学图像数据集,都只含有图像级别的分类标签,而有病灶级别标注的数据集,数量往往不足1000,而且大都针对单一类型的病灶,因此训练出的模型只能检测一种病灶。而实际病灶的种类非常之多,这种方式就显得有些低效。
2017年,同样由吕乐和罗纳德·撒莫斯博士率领的另一个NIH临床中心研究团队,发布了一个含有超过10万匿名胸部X光片的标注图像数据集,大幅推进了相关医疗AI研发,为医生提供了更好的临床诊断决策辅助。
这一次,吕乐、罗纳德·撒莫斯和他们在NIH的同事,朝着一个更加宏大的目标进发——构建一个囊括各种不同病灶的标注数据集,并在此基础上设计一个能够检测多种病灶的深度学习模型,从而更准确、自动地衡量患者体内所有病灶的大小,实现全身范围的癌症评估。
“美国的临床医疗数据库里,积累了医生在临床实践中留下的成千上万个病人的标注数据,”吕乐博士说:“但是,这些标注往往不完整,是医生为了辅助他们自己的工作进行的,不适合用于训练AI系统。”
而为了训练AI系统,请医生专门对大量的医疗图像进行标注又是不现实的,因为这需要耗费大量的人力和时间成本。因此,这件事情只有靠计算机设计深度学习算法来完成。
而要设计深度学习算法,关键就是带有准确标记的医疗图像大数据。去除隐私后的公共医疗大数据本就不多,而且不同于其他类型的数据标注,医疗图像标注需要丰富的临床经验,即使是资深医生,有时候也会在某个标注上产生意见分歧。
其结果就是,真正可用的标注医疗图像数据集稀缺,尽管各种医院诊所里积累了大量的医疗图像数据。
要设计深度学习算法解决临床问题,“需要对技术和临床实践两方面都拥有深刻的理解,”吕乐博士说:“AI 不是把放射科里面的东西再重新做一遍。哪些需求是临床上迫切需要的,哪些需求是当前技术能够满足的,将放射科学、计算机科学和人工智能结合在一起时,有很多需要深度考量和仔细权衡的东西,蛮干是不行的。”
为了构建DeepLesion数据集,研究人员倾注了一年多的时间,期间在国际医学图像计算与计算机辅助干预大会(MICCAI)、北美放射学年会(RSNA)等医学和放射影像国际顶会以及CVPR上,先后发表了12篇研究论文。
他们将工作成果汇聚为《DeepLesion: automated mining of large-scale lesion annotations anduniversal lesion detection with deep learning》,刊发在《Journalof Medical Imaging》。

现在,任何人都能访问并使用NIH的DeepLesion数据集(链接见文末),医生能够更好地进行自动临床病灶测量或者检索相似病例,AI研究人员则能进一步完善和推动AI诊疗技术和产品的研发。
接下来,NIH临床中心希望通过收集更多数据,不断完善DeepLesion,提高其检测的准确性。研究人员还考虑将现在仅含CT图像的DeepLesion进一步扩展,比如纳入MRI图像,并与其他医院的数据相结合,构建更加丰富的临床医疗图像库。
在NIH临床中心,每当有患者做完CT扫描后,相应的扫描图像都会被送至放射科医生处,然后医生会对图像进行解读。
接着,放射科医生会使用一种类似电子书签的工具,测量并标记CT扫描中具有临床意义的发现。与普通的实体书签类似,放射科医生会保存书签的位置,并将重要发现加上标记,以便以后能再次查阅。
这些书签的内容和形式很复杂,包括箭头、线条、长短径和文字说明等,用于标记病灶的确切位置和大小,以便专家识别病情发展或发现新的疾病。
在计算机视觉领域,为了收集图像标签,研究者会采用众包或搜索引擎抓取,但这并不适用于医学图像,因为医学图像的标注需要大量的医学知识和临床经验。
在开发DeepLesion数据集时,研究人员决定使用NIH临床PACS数据库中的“RECIST diameters”(Response Evaluation Criteria In Solid Tumors)长短径标注,这种标注是医生用来测量病灶大小的,被测量的一般都是医生认为比较重要的病灶,能够提供最精确的信息,从而确保数据的有效性。此外,这种类型的标注也是NIH数据库中所有标注类型中占比最高的一种。
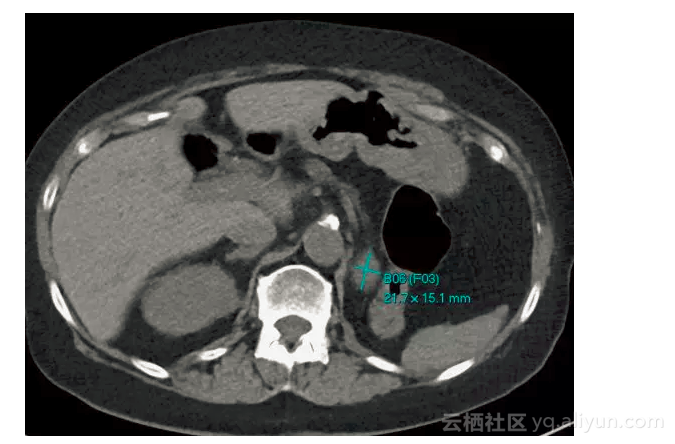
蓝线代表医生在日常工作中对病灶进行的长短径标注。来源: [1]
决定了要使用的标注类型后,DeepLesion项目的命名人、NIH临床中心的王潚崧博士与NIH PACS技术管理人员协商,确定了下载方案。随后团队编写程序,把含有长短径标注的图像、图像属性和标注从原始数据中提取出来,再按照病人编号、CT扫描编号(study index)、序列号(series number)和横断层编号(slice number)等进行整理。
在实际场景中,每个病人会进行多次CT扫描(每次扫描的时间点不同),每次扫描会生成多个序列(每个序列的造影剂增强、扫描重建参数等不同),而每个序列都是由几十到上百个横断层图像组成的3D影像。
数据清理和脱敏是一项浩大的工程。由于下载的电子书签中含有个别错误,研究人员设计了一些规则,清除掉这些错误,噪声、slice interval 信息的计算等,也都进行了手工纠正。
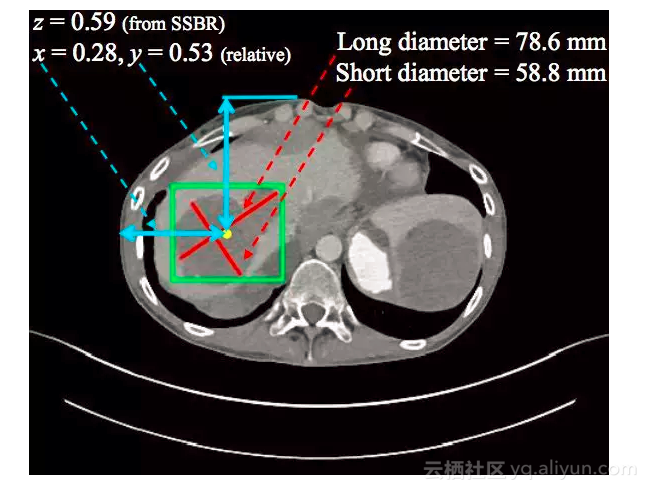
十字交叉的红线代表医生日常标注的长短径标注,绿框代表根据长短径计算出的边界框,黄点代表边界框的中心,两条相互垂直的蓝线表示病灶的 x 轴和 y 轴,z 轴根据SSBR[3]算出。来源:[3]
对于图像里可能含有的隐私信息,他们先用算法检测图像里的文字,然后人力检查了图像里是否有特别的首饰或者明显的人脸,最后手工抹除相关部分。
“CT图像的动态范围较大,因此我们采用16比特无损压缩png格式来保存所有图像。这些图像的使用方法可以参考数据集的FAQ文件,也可以参考论文[5]的源代码。”
“在开始这个项目之前,没想到构建数据库有这么复杂,”DeepLesion论文的第一作者、NIH临床中心的博士后研究员闫轲告诉新智元:“不过想到能给其他研究者带来好处,我们就有了持续前行的动力。”
构建通用疾病检测模型,推进精准个性化医疗
最终整理好的DeepLesion数据集,含有32735个带标记的病灶实例,包括来自全身各个部位的关键影像学发现,比如肺结节、肝肿瘤、淋巴结肿大等。
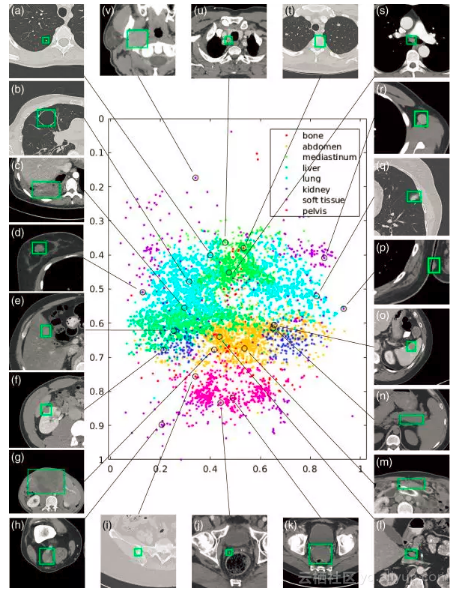
DeepLesion数据集中一个子集(15%)的可视化结果,包含骨骼、肝脏、肺部、腹部、软组织等全身各个部位的数据。散点图是按照病灶在身体中的相对位置绘制的,可以看做一个人体的正面照。来源:[1]
在实验过程中,研究人员意识到,虽然不同病灶的外观差异较大,但目前的深度学习算法完全有能力同时检测出各种病灶,从而提供了一个很有希望的新方向——创建一个具有统一框架的大规模通用病灶检测器。
这个通用检测器可以作为一种初步的检测工具来使用,如有需要再将其检测结果发送给在某些特定类型的病灶上训练过的其他专用检测模型。
闫轲说:“我们的数据集里包含了各种病灶,训练得到的是一个通用的检测器,虽然在特定种类的病灶上精度可能比不过专用的检测器,但优点在于覆盖了各种病灶,可以用来对病人的扫描影像进行一次性的初筛,降低漏检。”
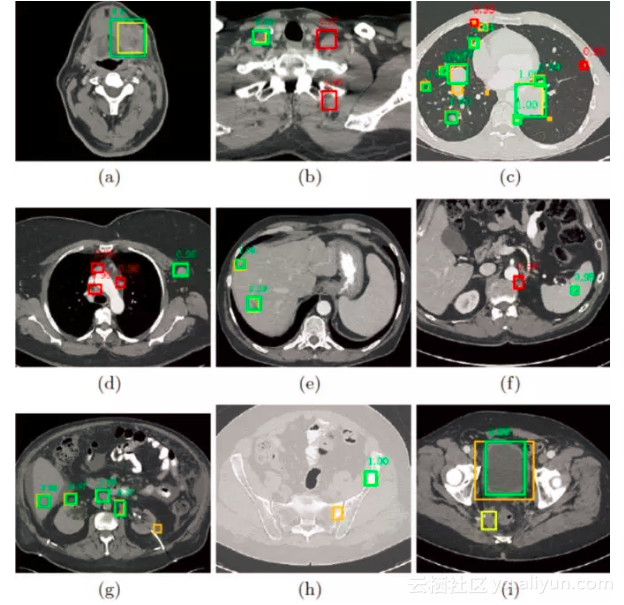
黄色和橙色框是ground-truth,绿色框是算法正确检测到的病灶,红色框是误检。算法可以较好地检测出各种病灶,但仍有一些漏检和误检。来源:论文[5]
通用病灶检测的难度远高于特定病灶检测,DeepLesion中包含肺、肝、肾、淋巴、胰腺、骨骼、软组织等各种病灶,病灶类内差异大,类间差异小(肺、肝的病灶相对容易检测一些,而一些腹腔中的病灶与周围正常组织差异较小)。继续收集数据,可以进一步提高准确率。另外,算法的改进空间也还有很大。
为了改进病灶检测的精度,NIH研究人员在MICCAI 2018上发表论文《3D Context Enhanced Region-based Convolutional Neural Network for End-to-End Lesion Detection》[5],提出了一种利用3D信息的检测算法,将病灶识别准确率提高到了84.37%(4 false positives per image)。算法代码见[5]。
除了自动检测病灶,DeepLesion还可以用于一些其他问题,比如对数据集中每个病灶进行测量。“病灶的测量是放射科医生的主要任务之一,”闫轲表示:“DeepLesion可以用于设计训练一个自动测量病灶的算法,减轻医生的负担,同时降低不同医生带来的个体差异。”
此外,这个数据集还可以用于进行病灶的分割、分类研究。NIH临床中心的蔡进铮博士和唐有宝博士利用DeepLesion,提出了弱监督病灶分割算法,相关论文《AccurateWeakly-Supervised Deep Lesion Segmentation using Large-Scale Clinical Annotations: Slice-Propagated 3D Mask Generation from 2D RECIST》发表在了今年的医学影像顶会MICCAI 2018。蔡进铮和唐有宝的工作还把所有的二维直径测量,用弱标注的深度学习算法,变成了高质量的肿瘤三维体积测量。
基于DeepLesion,提出的弱监督病灶分割算法得到北美放射科医生广泛好评。来源:Twitter
这项工作在北美放射科专科医生领域里引发了强烈反响,并收到了非常高的正面评价!主要原因是目前为止基于放射科医生人工标注的、在肿瘤图像最大横截面上的二维直径测量,也即前文提到的RECIST,是记算并比较肿瘤大小的现行临床标准。在实际工作中会造成很大的不决定性。基于绝对三维体积的肿瘤大小测量是公认的黄金标准,但因为制作费事费时,在临床中并没有被广泛应用。
这项工作基于DeepLesion数据库,提出了基于弱监督的深度学习肿瘤图像分割算法,从临床大数据中的直接利用原有的RECIST标注,无需任何额外的人工标注,即可学习和恢复三维的肿瘤分割和体积测量,并取得了好的定量和定性结果。扩展的期刊版本正在准备投稿《Radiology》。
“医疗AI技术的发展就是要让医生用起来有价值,真心爱用,”吕乐博士说:“Augmented intelligence for adding clinical value!”
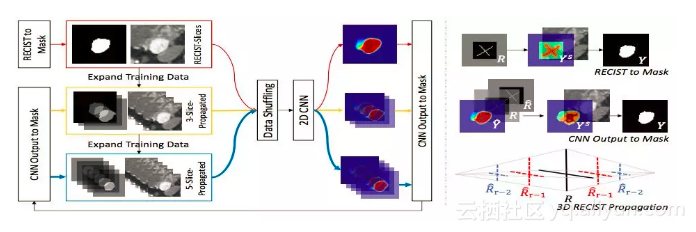
把所有的二维直径测量变成高质量的肿瘤三维体积测量
DeepLesion里大部分病人都有多次CT扫描的数据,而每次扫描又有多个不同病灶的标注,这为研究不同病灶之间的关系,以及同一病灶随时间的变化提供了可能。
“总之,这是一个给人很大想象和发挥空间的数据集。”闫轲说。
大规模的临床数据库是AI在临床真正落地的唯一路径;但标注几乎永远不可能是完美的,这就对新的更鲁棒的深度学习算法提出了更多以及更高的要求。
DeepLesion的获取方式也决定了它存在着一定的局限性。例如,医生在工作中通常只会标注有代表性的病灶,因此,数据集中存在着一些没有标出来的病灶。“我们也鼓励有余力的医生能够在DeepLesion上继续补全病灶标注,以造福广大研究者。”
DeepLesion 图像数据集下载(230G)
https://nihcc.box.com/v/DeepLesion
参考资料
[1] Ke Yan, Xiaosong Wang, Le Lu, Ronald M. Summers, “DeepLesion:automated mining of large-scale lesion annotations and universal lesiondetection with deep learning,” J. Med. Imag. 5(3), 036501 (2018), doi:10.1117/1.JMI.5.3.036501. (数据集构建和病灶检测)
[2] https://arxiv.org/abs/1707.03891(ISBI2017, body part regression)
[3] https://arxiv.org/abs/1711.10535 (CVPR 2018, Deep Lesion Graphs in the Wild: Relationship Learning and Organization of SignificantRadiology Image Findings in a Diverse Large-scale Lesion Database, 病灶检索与匹配)
[4] https://arxiv.org/abs/1807.01172 (MICCAI 2018; AccurateWeakly-Supervised Deep Lesion Segmentation using Large-Scale Clinical Annotations: Slice-Propagated 3D Mask Generation from 2D RECIST, 病灶分割)
[5] https://arxiv.org/abs/1806.09648(MICCAI 2018, 3DContext Enhanced Region-based Convolutional Neural Network for End-to-EndLesion Detection, 改进的病灶检测) ,源代码地址:https://github.com/rsummers11/CADLab/tree/master/lesion_detector_3DCE
[6] https://arxiv.org/abs/1806.09507 (MICCAI2018,Semi-Automatic RECIST Labeling on CT Scans with Cascaded Convolutional NeuralNetworks,病灶测量)
[7] https://arxiv.org/abs/1807.07144 (MLMI 2018; CT Image Enhancement Using Stacked Generative Adversarial Networks and Transfer Learningfor Lesion Segmentation Improvement,病灶图像增强)
[8] K Yan, Bethesda, MD;X Wang, PhD; L Lu, PhD; L Zhang; A P Harrison, PhD; M Bagheri, MD; et al. "Relationship Learning and Organization of Significant Radiology Image Findings for Lesion Retrieval and Matching"(RSNA ScientificPapers)
[9] Y Tang, PhD, Bethesda, MD; J Cai; L Lu, PhD; A P Harrison, PhD; K Yan; J Xiao; et al. "CT Image Enhancement for Lesion Segmentation Using Stacked Generative Adversarial Networks"(RSNA Scientific Posters)
[10] K Yan, Bethesda, MD; M Bagheri, MD; R M Summers, MD ,PhD; "3DContext Enhanced Region-based Convolutional Neural Network for Universal Lesion Detection in a Large Database of 32,735 Manually Measured Lesions on BodyCT"(RSNA Alternate)
[11] Youbao Tang, Adam P. Harrison, Mohammadhadi Bagheri, Jing Xiao, Ronald M. Summers. "Semi-Automatic RECIST Labeling on CT Scans withCascaded Convolutional Neural Networks" (RSNA Alternate)
[12] https://arxiv.org/abs/1710.01766 (initial version, RSNA 2017)
原文发布时间为:2018-08-15
本文作者:大明,闻菲
本文来自云栖社区合作伙伴新智元,了解相关信息可以关注“新智元”微信公众号
